Bioinformática - PhyML: alinhamento de sequências nucleotídicas em ambiente paralelo
O PhyML é um software utilizado para se fazer estimativa e probabilidade filogênica em alinhamento de sequências nucleotídicas ou sequências de anino ácido. Atualmente está na versão 3.0, seu desenvolvimento teve participação de diversos cientistas de algumas universidades no mundo.
[ Hits: 31.812 ]
Por: José Cleydson Ferreira da Silva em 27/07/2010
Sumário - Sobre o PhyML
Sumário
1. Sobre o PhyML2. Menus do PhyML
2.1 Submenu de entrada de dados
2.2 Submenu de modelos de substituição
2.3 submenu - Pesquisa de Árvore
3. Usando PhyML em linha de comando
4. Instalando PHYML em ambiente paralelizado
4.1Download do PHYML
5.0 Sobre o autor
6.0 Referências
1. Sobre o PhyML
O PhyML é um software utilizado para se fazer estimativa e probabilidade filogênica em alinhamento de sequências nucleotídicas ou sequências de anino acido. Atualmente está na versão 3.0, seu desenvolvimento teve participação de diversos cientistas de algumas universidades como apresenta o quadro abaixo: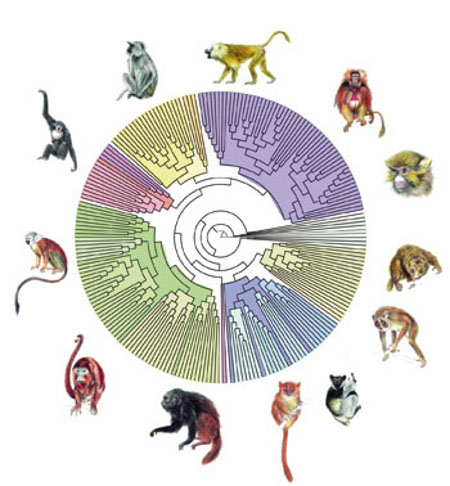
Departamento de Estatística. Universidade de Auckland. New Zealand
Universidade de Montpellier II - CNRS UMR 5506. LIRMM. Montpellier França.
e-mail : guindon@stat.auckland.ac.nz
Olivier Gascuel
Universidade Montpellier II - CNRS UMR 5506. LIRMM. Montpellier França.
e-mail : gascuel@lirmm.fr
Jean-Francois Dufayard
Universidade Montpellier II - CNRS UMR 5506. LIRMM. Montpellier França.
e-mail : jeanfrancois.dufayard@gmail.com
** Authors (before 2006)... **
Wim Hodrijk
Atualmente trabalhando na 'Rede nacional de Bioinformática - National Bioinformatics Network'. Cape Town, Africa do Sul
e-mail : wim@santafe.edu
Franck Lethiec
Universidade Montpellier II - CNRS UMR 5506. LIRMM. Montpellier França.
e-mail : lethiec@lirmm.fr
Este software é utilizado em linha de comando e possui uma interface simples com diversas diretivas de configuração e parâmetros que correspondem métodos estáticos, analise de sequência e entre outros.
Os métodos utilizado pelo PhyML dá-se também por Máxima Verosimilhança para reconstrução Filogenética, o que possibilita encontrar a árvore que mais provavelmente explicaria a evolução de determinado indivíduo . Em seguida podemos verificar os menus e suas funções.
2. Menus do PhyML
Os parâmetros de configuração podem ser utilizados de duas formas: Um por meio da interface do software e outro por meio de comandos shell. A interface do pode ser usada acessando menus conforme as letras que identificam as funções, a seguir podemos apresentá-las.2.1 Submenu de entrada de dados
[D] ............................... Data type (DNA/AA)Tipo de dados. Podendo ser DNA ou aminoácido.
[I] ...... Input sequences interleaved (or sequential)
Entrada de intercalada de sequência.
[M] ....................... Analyze multiple data sets
Analise múltipla de dados.
2.2 Submenu de modelos de substituição
[M] ................. Model of nucleotide substitutionModelo de substituição de Nucleotídios
[M] ................ Model of amino-acids substitution
Modelo de substituição de aminoácidos
[F] ................. Optimise equilibrium frequencies
Otimizar frequência
[F] . Amino acid frequencies (empirical/model defined)
Frequência de aminoácidos com modelo empírico definido
[T] .................... Ts/tv ratio (fixed/estimated)
Fixar ou estimar transcrição / transversão com relação a Máxima Verosimilhança
[V] . Proportion of invariable sites (fixed/estimated)
Proporção de viabilidade fixa ou estimada
[R] ....... One category of substitution rate (yes/no)
Categoria de substituição de taxa
[C] ........... Number of substitution rate categories
Numero de substituição da categoria taxas
2.3 submenu - Pesquisa de Árvore
[O] ........................... Optimise tree topologyOtimizar topologia da árvore
[S] .................. Tree topology search operations
Operações de pesquisa na topologia da árvore
[R] ......................... Use random starting tree
Iniciar árvore usando aleatoriedade
[N] .................. Number of random starting trees
Iniciar árvore com numero aleatório
[U] ..................... Input tree (BioNJ/user tree)
Entrar com uma árvore
2.4 Suporte a Brunch
[B] ................ Non parametric bootstrap analysisSem analise para métrica bootstrap
[A] ................ Approximate likelihood ratio test
Teste de aproximação Verosimilhança.
2. Usando PhyML em linha de comando
3. Instalando PHYML em ambiente paralelizado
4. Sobre autor
Acessando desktop remotamente com noMachine
Bing: Medindo velocidade da conexão no Linux
Cairo-Dock - Seu desktop Linux com cara de MAC
Bioinformática - Instalação do SNAP workbench
Instalando o MEO Cloud em qualquer distro GNU/Linux
SIAGES: Uma oportunidade de negócio com software livre
Usando temas aleatórios no GDM
Grande Kuruma!!
Parabéns pelo artigo. Apesar de eu não estar familiarizado com coisas como:
probabilidade filogênica
alinhamento de sequências nucleotídicas
sequências de anino ácido
Eu acho que isso irá e muito ajudar a comunidade do software livre.
E mais do que nunca
VIDA LONGA AO SL!!
Dailson Fernandes
http://www.dailson.com.br
Olá Dailson,
Grande Guerreiro, obrigado pelo incentivo meu camarada. Vou começar uma serie longa desses artigos, estou pensando em enviar um trabalho para o VOL DAY, em Bebedouro-SP, mas estou pensando se poderá ser aceito ou não. Esses artigos com certeza deram muito trabalho para validar a prática. Mas com certeza resolverá a vida dos Biologos e dos Bioinformatas.
Um abraço!
Patrocínio
Destaques
Artigos
Instalação Completa e Configuração Básica do Void Linux
A Fundação da Confiança Digital: A Importância Estratégica de uma PKI CA na Segurança de Dados
Como enviar dicas ou artigos para o Viva o Linux
Como Ativar a Aceleração por GPU (ROCm) no Ollama para AMD Navi 10 (RX 5700 XT / 5600) no Gentoo
Dicas
Samba 4 AD-DC 2026: Backup & Restore
Samba 4 AD-DC 2026: LAPS (Randomizando senhas locais).
Cairo Dock ainda funcional nos dias de hoje
Configuração de IP fixo via nmcli e resolução de nomes via /etc/hosts no Gentoo
Tópicos
Top 10 do mês
-

Xerxes
1° lugar - 128.857 pts -

Fábio Berbert de Paula
2° lugar - 55.587 pts -

Buckminster
3° lugar - 34.328 pts -

Sidnei Serra
4° lugar - 20.482 pts -
Alberto Federman Neto.
5° lugar - 20.535 pts -

Mauricio Ferrari (LinuxProativo)
6° lugar - 18.767 pts -

Daniel Lara Souza
7° lugar - 18.512 pts -

Alessandro de Oliveira Faria (A.K.A. CABELO)
8° lugar - 17.934 pts -

edps
9° lugar - 16.846 pts -

Andre (pinduvoz)
10° lugar - 14.420 pts




