Bioinformática - Clustalw-MPI: Análise Filogenética utilizando computação paralela e distribuída
O software Clustalw-MPI é a ferramenta mais popular na implementação de múltiplo alinhamento de sequência de DNA. Sua principal diferença entre o software ClustalX dá-se a que ele possui uma melhor performance computacional devido realização do processamento em paralelo ou distribuído.
[ Hits: 21.336 ]
Por: José Cleydson Ferreira da Silva em 09/07/2010
Instalação do Clustalw-MPI
Sobre Clustalw-MPI
O software Clustalw-MPI é a ferramenta mais popular na implementação de múltiplo alinhamento de sequência de DNA. Sua principal diferença entre o software ClustalX dá-se a que ele possui uma melhor performance computacional devido realização do processamento em paralelo ou distribuído.A biblioteca utilizada na divisão do esforço computacional é chamada de MPI (Mensage Passing Interface). Ela proporciona redução no tempo de execução de determinadas processos.
O alinhamento pode ser obtido através de três estados:
- Alinhamento Pairwise
- Geração da arvore guia
- Alinhamento progressivo
O Clustalw-MPI é software semi-livre segundo a sua forma de licenciamento. Embora possua características de software livre como permissão de execução de programa, de estudo de seu funcionamento, de liberdade de redistribuição e de modificação, não há permissão de uso para fins comerciais. O autor do software chama-se Dr. Kuo-Bin Li <kbli@ym.edu.tw>, do instituto de bioinformática de Singapura.
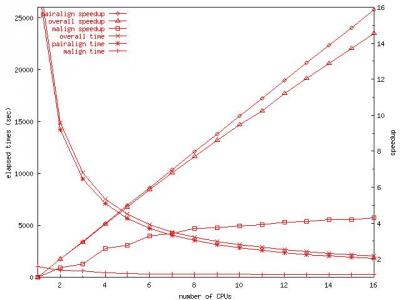
Performance Benchmark
Instalação do Clustalw-MPI
A instalação pode ser realizada de duas formas, através do gerenciador de pacotes da distribuição Debian ou derivados ou compilando através de código fonte. Segue abaixo as duas formas de instalação.Instalação via gerenciador de pacotes:
# apt-get install clustalw-mpi
Para se fazer a instalação por meio do código fonte é necessário verificar a configuração do arquivo Makefile. Os parâmetros de bem configurados podem proporcionar um bom funcionamento do software.
Parâmetros de configuração:
- CC = mpicc
- CFLAGS = -c -g
- or
- CFLAGS = -c -O3
Pode-se obter o código fonte do software no site do Instituto de Bioinformática de Singapura. Siga o processo de instalação por meio do código fonte no quadro abaixo.
Download via terminal:
# wget http://www.bii.a-star.edu.sg/docs/software/clustalw-mpi-0.13.tar.gz
Descompactar o arquivo:
# clustalw-mpi-0.13.tar.gz
Compilar o software:
# cd clustalw-mpi-0.13
# make
Um arquivo binário será gerado no diretório corrente um arquivo com o nome clustalw-mpi, entretanto pode-se movê-lo ou copiá-lo para o diretório /usr/bin para o que shell o identifique como um comando.
# mv clustalw-mpi /usr/bin/clustalw-mpi
ou
# cp clustalw-mpi /usr/bin/clustalw-mpi
Para se ter acesso ao executável que acabou de ser compilado, use o comando clustalw-mpi no terminal.
Usando clustalw-mpi
A usabilidade desse software dá-se por linhas de comando ou prompt de comando, a sintaxe da linha de comando determina usar os seguintes atributos.| mpirun | Executa tarefas paralelas em MPI. |
| -np | -npersocket, número de processos para cada nó. |
| 5 | Número de processadores. |
| clustalw-mp | Software que realiza o alinhamento de múltiplas sequências. |
| -Infile | Arquivos de entrada no formato FASTA. |
| -newtree | Arquivo de saída para gerar a árvore. |
Há 3 situações que caracterizam a usabilidade do Clustalw-MPI, que consiste em fazer alinhamento completo de múltiplas sequências, construir apenas árvore guia, múltiplo alinhamento de uma árvore existente e por fim a definição da variável de ambiente.
Alinhamento completo de múltiplas sequências
O alinhamento completo de múltiplas sequências usará um nó para mestre que por sua vez, irá gerenciar os processos enviados para os outros nós que se encontrem envolvidos no esforço de realização da determinada tarefa em paralelo.Usando por exemplo 10 processadores ou nós em rede, um será o gerenciador de envio e recebimento do resultado do processo e os outros 9 processadores ou nós serão utilizados para realizar os processamento da tarefa. A sintaxe de usabilidade desse contexto está representada.
# mpirun -cc 10 ./clustalw-mpi -infile=dele.input
# mpirun -np 10 ./clustalw-mpi -infile=CFTR.input
Árvore guia
Esse contexto refere-se apenas na criação de árvores, por meio de um arquivo de sequências. A sintaxe requer a indicação do arquivo de sequência e a definição de um novo arquivo onde será construída a arvore.# mpirun -np 5 ./clustalw-mpi -infile=dele.input -newtree=dele.mytree
# mpirun -np 5 ./clustalw-mpi -infile=CFTR.input -newtree=CFTR.mytree
Múltiplo alinhamento de uma árvore existente
Pode-se usar uma árvore já existente e fazer o alinhamento dela, para que isso ocorra é necessário usar o parâmetro -usetree.# mpirun -np 5 ./clustalw-mpi -infile=dele.input -usetree=dele.mytree
# mpirun -np 5 ./clustalw-mpi -infile=CFTR.input -usetree=CFTR.mytree
Variável de ambiente
A variável de ambiente a ser definida chama-se CLUSTALG_PARALLEL_PDIFF. A mesma é utilizada para executar alinhamento progressivo baseado em um pdiff paralelizado. Por padrão essa variável se encontra em estado não definida e o alinhamento progressivo será paralelizado conforme a estrutura da arvore de neighbor-joining.Habilitar essa variável requer entender a respeito de algumas funções, bem como parallelized pdiff(), prfalign(), e no entanto o manual do software não menciona como definir a variável CLUSTALG_PARALLEL_PDIFF. Deixá-la desabilitada não irá afetar a usabilidade e o desempenho do software.
2. Sobre o autor
Gerência de projetos com Redmine
Como migrar banco de dados MySQL para PostgreSQL
Compiz - Conhecendo a fundo II
Elaborando vídeo-aula no Linux com Gtk-recordMydesktop
Seu Slackware atualizado com SWARET
Rastrear e recuperar seu PC ou notebook roubado
Administração de sistema via WEB
Garantindo o funcionamento de serviços com o restartd
GRIVE - Google Drive Linux (Grátis)
Cara, estou adorando essa serie de postagens sobre bioinformática. Sempre senti falta de alguma coisa no gênero em nossa lingua.
Muito bom!
Olá Diniz,
Realmente é uma boa oportunidade de apresentar essa série inedita de artigos em bioinformática. Espero postar outros artigos e apresentar outros softwares que fazem montagem de genona. Aproveite para indica-los a outros bioinformatas.
Obrigado, um abraço!
Olá Mauricio ('mecantao '),
O artigo realmente foi bem elaborado. Esse é um projeto que estou iniciando ainda em graduação, documentar grande parte dos softwares da área de bioinformática que são livres e de código aberto. Sempre dou preferência para aqueles que fazem processamento paralelo, o tempo gasto em analises de DNA são imensos e o paralelismo nos ajuda a diminuir esse tempo. Há outros artigos que escrevi e também uma comunidade aqui no viva o linux para tirar-mos dúvidas e postar-mos algumas novidades. Se estiver interessando entre na comunidade Open Source Bioinformatics.
Um abraço.
Patrocínio
Destaques
Artigos
A Fundação da Confiança Digital: A Importância Estratégica de uma PKI CA na Segurança de Dados
Como enviar dicas ou artigos para o Viva o Linux
Como Ativar a Aceleração por GPU (ROCm) no Ollama para AMD Navi 10 (RX 5700 XT / 5600) no Gentoo
Dicas
Samba 4 AD-DC 2026: LAPS (Randomizando senhas locais).
Cairo Dock ainda funcional nos dias de hoje
Configuração de IP fixo via nmcli e resolução de nomes via /etc/hosts no Gentoo
Removendo o bloqueio por erros de senha no Gentoo (systemd)
Papel de Parede Animado no KDE Plasma 6 (Com dicas para Gentoo)
Tópicos
Top 10 do mês
-

Xerxes
1° lugar - 128.774 pts -

Fábio Berbert de Paula
2° lugar - 55.495 pts -

Buckminster
3° lugar - 30.704 pts -

Sidnei Serra
4° lugar - 21.641 pts -
Alberto Federman Neto.
5° lugar - 19.682 pts -

Daniel Lara Souza
6° lugar - 19.425 pts -

Mauricio Ferrari (LinuxProativo)
7° lugar - 18.797 pts -

Alessandro de Oliveira Faria (A.K.A. CABELO)
8° lugar - 17.697 pts -

edps
9° lugar - 16.576 pts -

Diego Mendes Rodrigues
10° lugar - 14.127 pts